Arbeitsgruppe Digitale Pathologie
Projekt "Virtuelle Mikroskopie"
Leitung: apl. Prof. Dr. med. Thomas Kalinski
Ziel dieses Projektes ist die Einführung, Evaluierung und Etablierung der virtuellen Mikroskopie in der pathologischen Lehre, Forschung und Diagnostik.
Virtuelle mikroskopische Präparate entsprechen vollständigen digitalen Abbildern konventioneller mikroskopischer Präparate. Die Darstellung erfolgt am Computerbildschirm. Zur Digitalisierung wird ein Scanner eingesetzt, der virtuelle mikroskopische Präparate in einer dem konventionellen Lichtmikroskop entsprechenden Auflösung erstellt. Die Dateien werden auf einem Server gespeichert und stehen so jederzeit zur Verfügung.
Virtuelle mikroskopische Präparate sind über das Internet unabhängig vom Ort jederzeit verfügbar. Ein Mikroskop ist nicht erforderlich. Konsultationsmeinungen können mit Hilfe dieser Technik ohne Zeitverlust durch das Versenden von Präparaten weltweit eingeholt oder abgegeben werden (sog. Telepathologie). Darüber hinaus können virtuelle Präparate nicht altern und ohne Qualitätsverlust beliebig oft kopiert werden.
Die Herausforderung besteht in der Organisation der Daten und der Integration in eine moderne Instituts- und Klinik-übergreifende Systeminfrastruktur. Am Universitätsklinikum Magdeburg ist das klinikweite Bildarchivierungs- und Kommunikationssytem PACS (picture archiving and communication system) im Einsatz, das den DICOM-Standard (digital imaging and communications in medicine) erfüllt. Durch die Einbindung der virtuellen Mikroskopie in das Informationssystem der Pathologie (IS-P), das Befunde und Daten in elektronischen Patientenakten zur Verfügung stellt, entsteht die Digitale Pathologie.
Kooperationspartner
- Institut für Biometrie und Medizinische Informatik, Otto-von-Guericke-Universität Magdeburg (Prof. Dr. rer. nat. Dr. med. J. Bernarding)
- Medizinisches Rechenzentrum, Universitätsmedizin Magdeburg (Dr.-Ing. R. Waschipky)
Weiterführende Informationen & Kontakt
Projekt "Multispektrales Imaging"
Leitung: PD Dr. rer. nat. habil. Norbert Naß, Prof. Dr. med. univ. Dr. sc. nat. Johannes Haybäck
Mitarbeiterinnen: Hella Wolf, Kerstin Werner
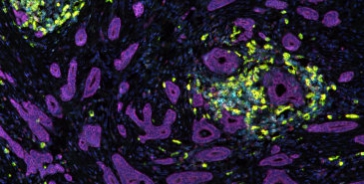
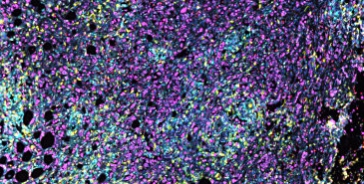
Das diagnostische Routine-Verfahren der Immunhistochemie ermöglicht jeweils die Darstellung eines einzelnen Biomarkers pro Gewebeschnitt. Für zusätzliche Biomarker werden weitere Schnitte benötigt, die dann allerdings andere Zellen aus einer veränderten Schnittebene enthalten. Einige Zelltypen können aber nur durch Mehrfachfärbungen voneinander unterschieden werden. Beim multispektralen Imaging kann mit Hilfe von mehreren Fluoreszenzfarbstoffen, kombiniert mit einer spektralen Analyse dieser Farbstoffe, die Anzahl der gleichzeitig untersuchten Biomarker auf derzeit sieben erweitert werden. Damit können zum Beispiel die Tumor-infiltrierenden Lymphozyten, die wichtig für die Immuntherapie sind, in einem Schnitt identifiziert und damit auch ihre räumliche Zuordnung untereinander und zum Tumor untersucht werden.
Die Bilder zeigen Färbungen von Tumor-infiltrierenden Lymphozyten (TILs) in Mammakarzinomen der Grade 1 und 3. Die Färbung ist eine Kombination von CD4, CD8, CD20, CD45Ro, DAPI, FOXP3 und Panzytokeratin.
Routine pathological diagnostic often relies on the detection of a single biomarker per tissue slice by immunohistochemistry. However, identification of several cell types, especially immune cells, requires the detection of more several biomarkers at once. This can be achieved by applying a number of spectrally differing fluorescent dyes in a technique called multispectral imaging, which currently allows the identification of up to seven biomarkers on a single slide. We are currently applying this technique for the detection and characterization of tumor infiltrating lymphocytes. Ongoing research shows that the presence and spatial arrangement of such cells can have great impact on the efficacy of immunotherapy in cancer. |
| Kontakt: PD Dr. rer. nat. habil. Norbert Naß, Telefon 0391-67-17876 | 0391-67-17863 |
| norbert.nass@med.ovgu.de |